El Método 7×7
La resurrección de CRISPR
Para hablar de ello, un viejo amigo del programa, Lluís Montoliu, autor del paper e investigador del Centro Nacional de Biotecnología del CSIC (CNB-CSIC) y del CIBERER.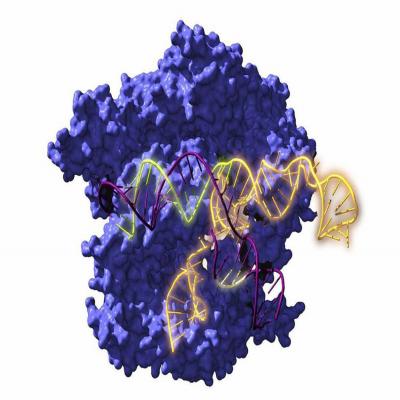
Aunque todavía no seamos conscientes de ello, vivimos en el inicio de una era revolucionaria: la era CRISPR. Estas siglas, acuñadas por el científico español Francis Mojica, describen uno de los elementos del sistemas inmunitario de bacterias y arqueas con el que, años después, Jennifer Doudna y Emmanuelle Charpentier idearon una poderosísima herramientas biotecnológica que permite cortar y pegar trozos de ADN con una facilidad pasmosa. Su herramienta les valió un Premio Nobel en Química poco tiempo después.
Hoy tratamos un episodio dentro de la saga CRISPR en el que un grupo de científicos españoles ha "resucitado" los ancestros de los CRISPR actuales... y no lo han hecho solo porque pueden, porque el conocimiento por el puro conocimiento es valioso sino porque se pueden derivar interesantísimas aplicaciones de ese viaje en el tiempo genético.
Para hablar de ello, un viejo amigo del programa, Lluís Montoliu, autor del paper e investigador del Centro Nacional de Biotecnología del CSIC (CNB-CSIC) y del CIBERER.
Si queréis oler más, pasaos por SINC https://www.agenciasinc.es/Noticias/Resucitan-ancestros-de-las-tijeras-geneticas-CRISPR-de-hace-2.600-millones-de-anos
Este contenido es gratis y sólo te pido que, si te ha gustado, entretenido, iluminado de algún modo, lo compartas en tus redes y nos valores en tu plataforma de pódcast favorita. Gracias ;)
Publicado: 6 enero 2023
Transcripción
Antigüedad predicha, ahora entraremos en las migas de todo esto. El artículo aparece en la revista Nature Microbiology y si os apetece leer una pieza sobre el tema en profundidad, os animo a pasaros por la web de Zinc. Allí encontraréis un artículo que ha firmado la redacción sobre este tema. Pero como es un asunto que me gusta, sigo desde hace tiempo y conozco a algunos de los protagonistas, en este caso, incluso del paper en particular, pues he llamado al buen amigo Lluís Montoliu, investigador del
Centro Nacional de Biotecnología del CSIC, en el CNB CSIC y del CiberAir, y hemos hablado un ratito por teléfono, porque lo he pillado de vacaciones todavía, o de supuestas vacaciones, ya veis que atiendan los medios siempre, para que me contara no sólo qué han hecho en este trabajo, en este paper, sino también algunas de las cuestiones algo más teóricas que, vamos, me parecían interesantes y creo que han dado para una conversación, espero, también interesante.
Ya sabes, si esto te gusta o te interesa, pues compártelo, así lo agradeces. Os dejo con la conversación con Lluís y os aviso otra vez de esa calidad de conversación telefónica. Hasta ahora. Pues nada, Lluís, primero, enhorabuena, porque es un paper maravilloso y está teniendo un impacto la verdad, tremendo, ¿no? ¡Qué bien! Sí, yo creo que también ayuda las fechas, ¿no? Cuando nos dijeron que finalmente se iba a liberar el artículo a principios de año,
al principio nos quedamos un poquito así, que dijimos, madre mía, en medio de las fiestas, la Semana de Reyes, pero bueno, al final, luego lo resulta es que tampoco, a veces hay tantas noticias científicas en estos días, de repente una que además es singular y tiene también sus derivadas así, que te permiten especular, porque estamos hablando de hace miles de millones de años, pues yo creo que todo habrá sumado para que hayamos tenido
este impacto mediático, que os tenemos que agradecer a vosotros, a los medios. Para quien no se haya leído el paper, ¿cuál es tu resumen tweet de lo que habéis conseguido? Pues lo que hemos conseguido es reconstruir unas proteínas, unas nucleasas K, que son una parte de los sistemas CRISPR de edición genética, de bacterias que debieron vivir hace decenas, centenares o miles de millones de años.
Para sentar un poco el ABC, las bases de lo que alguien necesita comprender para valorar este paper. Uno, estamos hablando de algo que creo que empieza a sonar a mucha gente, tal vez no tanto como la PCR después de la pandemia, pero poco a poco, CRISPR suena mucho. CRISPR es, de manera muy simplificada, el sistema inmunitario que tienen algunas bacterias y arqueas. En general, la mayoría de bacterias y arqueas que lo usan para defenderse de los virus.
Básicamente es un sistema para reconocerlos, para quedarse con un fragmento de ellos, así como una fotografía, y la próxima vez que ese virus intenta volver a infectar la bacteria, la bacteria se acuerda de que este es un invasor y le monta una respuesta que acaba degradando el genoma del virus. Entonces es un sistema muy efectivo de base genética para luchar contra los virus. Y como para funcionar, lo que hace es, por un lado,
necesita detectar la secuencia genética de estos virus, porque no detecta las proteínas de la cápside, sino la secuencia genética de este atacante. Tiene que detectarlo y una vez detectarlo, como tú dices, algo que lo degrade, lo rompa, lo cambie, lo inutilice. Y esas son las bases que se han usado posteriormente. Estas son las bases que sentó nuestro colega Francisco, el microbiólogo de la Universidad de Alicante, hay que recordarlo siempre.
Él empezó a trabajar en estos temas hace exactamente 30 años, en el 93, y en el año 2003 se dio cuenta de que esto era, tal y como lo has contado, un sistema inmunitario, un sistema de defensa que tienen las bacterias. Era una cosa tan revolucionaria que le costó casi 3 años publicarlo. Por eso tenemos que citar su artículo que se acabó publicando en 2005, pero él el descubrimiento lo hizo en el 2003, 10 años después de empezar a trabajar.
En el 2005, cuando lo publicó, acabó publicándolo en una revista que no era de las primeras, pero que era una revista muy digna y que acabó siendo leído ese artículo por dos investigadoras, por Charpentier y por Dauna, que 7 años después, en 2012, se les ocurrió dar una vuelta a la tortilla al asunto y convertir ese sistema de defensa en un sistema de ericción, de recorta, pegue y colorea de nuestro ADN,
de cualquier ADN de cualquier ser vivo. Esto fue en el 2012 y 8 años después ellas recibieron muy merecidamente el Premio Nobel de Química. Ahora estamos en las primeras fases de una verdadera revolución, en el sentido de, creo que era Brenner quien decía que técnicas, técnicas, técnicas, que así es como se avanza en ciencia. CRISPR permite hacer nuevas preguntas y, sobre todo, intentar buscar nuevas maneras
de responder a preguntas que antes solo eran teóricas o imaginables, pero no ejecutables. Puedes hacer unos experimentos que anteriormente ni tan siquiera te los podías plantear. Modificar el ADN, pues lo sabemos hacer desde hace bastantes años, en particular en mamíferos como nosotros, lo sabemos hacer desde el año 1980. Pues imagínate, llevamos más de 40 años. Pero modificarlo con la precisión que requiere la corrección de un gen que es el causante
de la enfermedad, que a veces el origen del problema es apenas una sola letra del genoma que está mal puesta o que está eliminada o que está duplicada, modificarlo con ese nivel de precisión era algo que no podíamos hacer sencillamente. Y esto es algo que durante miles de millones de años los sistemas CRISPR han aprendido a hacer. Y, de nuevo, es un ejemplo más que nos recuerda que si tenemos que aprender de alguien,
pues tenemos que aprender de las bacterias, que nos llevan una ventaja considerable sobre el planeta. Ellos llevan miles de millones de años viviendo aquí, han tenido tiempo para probar y para hacer test de prácticamente cualquier idea que pudiera desarrollar la evolución y básicamente lo que nos llega a nosotros son sistemas ultra optimizados, súper purificados, sistemas que cuando los trasladamos
fuera de las bacterias y los pretendemos usar, pues acaban dándonos tantas alegrías como esta. Y, sin embargo, aquí hay una cierta ironía que tiene mucha miga y es que el trabajo que vosotros presentáis de hecho va a perseguir los modelos más antiguos, los menos actualizados, los menos pulidos y optimizados, pero porque eso tiene utilidad también. Y eso tiene una explicación porque a veces uno hay que ir, el diablo está en los detalles,
se dice, y a veces uno tiene que ir a lo que es la base del asunto para entenderlo. Aquí estamos todos súper contentos y revolucionados con las herramientas CRISPR, nosotros mismos en el laboratorio nos ha cambiado la vida, pero si levantamos la alfombra, en realidad, pues prácticamente todos los laboratorios estamos usando los sistemas CRISPR de una sola bacteria o de dos.
Y estas bacterias, cuyos sistemas CRISPR son tan útiles, resulta que son patógenas para nosotros. Estoy refiriendo a Streptococcus piogenes y a Staphylococcus aureus. Algunas de ellas quizá les suenerán a quienes los estén escuchando. El que se haya sacado el certificado de manipulación alimentaria, Staphylococcus aureus es un básico. Exactamente.
Son bacterias que causan infecciones. Piógenes, por ejemplo, causa otitis o erigitis. Entonces, ¿qué quiere decir esto? ¿Cuál es la consecuencia? Pues la consecuencia es que nuestro sistema inmunitario habitualmente va a conocer estas bacterias, habrá montado respuestas inmunes contra él, tendremos anticuerpos contra las bacterias y contra sus componentes, también contra sus componentes CRISPR. Entonces, si pretendemos usar alguna de estas proteínas Cas de CRISPR-Cas para tratar a un paciente
y resulta que el paciente tiene anticuerpos antiesas por CRISPR-Cas, el sistema inmunitario se encargará de eliminarlas y de atacarlas. Con lo cual, ahora le hemos hecho un pan con una torta. O sea, realmente no vamos a poder utilizarla. De ahí que, desde hace ya algunos años, muchos investigadores se han preocupado de intentar descubrir sistemas CRISPR lo más alejados posibles de las poblaciones humanas.
Y se han ido a los lugares más recónditos del planeta. Se han ido a Yellowstone, a los geysers, se han ido a la Antártida, se han ido a las fosas oceánicas de las Marianas, se han ido a la cima del Everest, a los volcanes, para buscar bacterias que prácticamente viven en prácticamente todos los lugares del mundo, incluso aquí en Huelva, en Río Tinto, que son ambientes muy extremos y que, con la esperanza de encontrar bacterias cuyos sistemas CRISPR sean lo suficientemente distintos
para que nuestro sistema inmunitario no los detecte. Bueno, pues nosotros hemos optado por una estrategia alternativa, igual de válida creemos, que es, en lugar de coger las maletas y viajar geográficamente, pues hacer un regreso al futuro y viajar en el tiempo. Hemos empezado con un montón de bacterias actuales. Hemos obtenido de cada una de ellas el gen que codifica para esta nucleasa, para esta nucleasa Cas9, que es la más característica de los sistemas CRISPR,
y dado que están más o menos evolutivamente relacionadas, se las hemos dado a un superordenador y le hemos dicho que nos busque los ancestros. Es algo así como hacer una recolección de bisnietos y decirle al ordenador búscame cuál es el bisabuelo que es común a todas ellas. Bueno, no solamente nos hemos quedado con el bisabuelo, nos hemos ido mucho más atrás. Se puede esto calibrar en el tiempo y nos hemos detenido pues en 37, 137, 200, 1000 y 2600 millones de años.
Esto es como la primera vez que nosotros oímos de esta propuesta. A mí me explotó la cabeza porque dije, madre mía, esto es una cantidad de años impresionante. La primera pregunta que se nos ocurre es, ¿pero acaso no podemos acudir al registro fósil? Bueno, es que no hay registro.
De hecho, tenemos que recordar que hace muy poco se ha identificado el adendo más antiguo y el adendo más antiguo tiene apenas dos millones de años. Y dos millones de años, que es una pasada de años, pues es prácticamente nada con la historia del planeta. En la Tierra se cree que tiene 4.500 millones de años. Claro, dos millones no es nada y la única manera de acudir a estas edades tan antiguas es hacer este recorrido inverso, este camino inverso y decirle al ordenador
que nos vaya calculando y nos vaya haciendo predicciones de proteínas que debieron existir, que podrían haber existido y que son compatibles con la variabilidad de proteínas actuales. ¿Tenemos claro que esas proteínas existieron? Pues no, no lo podemos tener claro. Pero sí que podemos aseverar que esas proteínas son compatibles con las que conocemos hoy en día. Y Luis, una vez tenéis este retrato robot que os hace la computadora,
digamos, extrapolando los ancestros de las proteínas que se conocen hoy, ¿cómo es? ¿Qué cara tiene? ¿Cuáles son las características que saltan? Básicamente el ordenador lo que nos da son secuencias de las proteínas que están formadas de aminoácidos. Tenemos 20 aminoácidos que se van repitiendo en diferentes combinaciones y esto hace que tengamos proteínas distintas. Entonces el ordenador nos dice, hace mil millones de años,
una casa nucleasa podría tener este aspecto y nos da la secuencia de aminoácidos. Y esta secuencia que nos la da escrita en un papel, pues se la podemos enviar a una empresa, que hay empresas hoy en día que básicamente a partir de esa secuencia te reconstruyen la proteína. O incluso puedes deducir cuál sería el ADN que las codifica. Sabemos cuál es el código genético, cada tres letras es un aminoácido,
pues podemos reconstruir el ADN y también podemos encargar la síntesis de ese ADN. En definitiva, convertimos una propuesta informática en una realidad. Y esa realidad es la que se la damos, lo que hicimos nosotros, a unas células humanas en cultivo y le preguntamos, ¿tú eres capaz de funcionar como una herramienta de edición? Y para nuestra sorpresa la respuesta es que sí. Y la respuesta es que sí con un grado de efectividad nada deseñable.
Realmente las más, entre comillas, jóvenes, las que apenas tienen 37 millones de años, pues funcionan prácticamente de forma muy similar a las actuales. Para que nos centremos, 37 millones de años, los dinosaurios desaparecen hace 65 o 66 millones de años. El primer dinosaurio que sale en la película Jurassic Park es este enorme barquiosaurio, el del cuello largo y la cola larga, ese desapareció hace 150 millones de años.
Pero es que nos vamos hasta 1.000 millones de años, que estos antes de lo que se llama la explosión cámbrica, que ocurrió hace 580, 600 millones de años, que dio lugar a prácticamente todos los diferentes animales que existen hoy en día. Con lo cual nos hemos ido realmente muy atrás. ¿Qué características tienen estas proteínas que las diferencian de las de ahora? Más allá de lo que tú decías, de que no tengan esta relación con especies que son tradicionalmente patogénicas,
pero en su funcionamiento, ¿tienen más piezas, menos piezas, alguna cosa que salte? Lo primero que vemos es que su capacidad para editar genéticamente disminuye a medida que nos vamos alejando. Es decir, la de 37 millones de años, que es la más joven que hemos reconstruido, funciona estupendamente. La de 137 funciona un poco peor, la de 200 un poco peor, la de 1.000 un poco peor. Y la de 2.600 es la que tiene un funcionamiento más precario.
Entonces uno se preguntaba, si no funciona, ¿para qué la habéis reconstruido? Pues resulta que esa que aparentemente no funciona tal y como esperamos o esperaríamos que funcione, resulta que tiene estas características singulares. Cuando empezamos a ver que no funcionaba tan bien para hacer la edición genética, nos preguntamos, oye, vamos a ver si son capaces de detectar diferentes tipos de secuencias,
o sea, con una menor especificidad. Y efectivamente esto es así. La especificidad de detección, lo específicas que son, va disminuyendo a medida que nos alejamos. Y esto también encaja con esa idea de que evidentemente hace miles de millones de años necesariamente los seres vivos tenían que ser más simples y la variabilidad y la variedad de seres vivos tenía que ser menor. Seguramente habría muchos menos virus que en la actualidad,
por lo tanto los requisitos para ser extraordinariamente específicas no debían ser tan importantes. Y efectivamente esto es lo que ocurre. Ahora en la actualidad las bacterias tienen que luchar contra millones de virus y entonces necesitan ser muy precisas, por eso los sistemas CRISPR actuales son tan, tan y tan precisos. Y la otra derivada que encontramos es que son capaces de cortar unas especies de ácidos nucleicos
distintas de las actuales. Las actuales en general cortan el ADN de doble cadena. También las hay que corten ADN de cadena sencilla o que corten ARN, que como sabemos también tiene una sola cadena, también las hay. Pero estas son las menos, mientras que cuando nos alejamos en el tiempo resulta que esas tan antiguas prefieren cortar moléculas de ácidos nucleicos de una sola cadena, que de nuevo vuelve a encajar con esa idea que tenemos de un mundo primitivo,
un mundo ARN, un mundo de moléculas de ácido nucleico de una sola cadena que debió preceder al que conocemos actualmente de ADN de doble cadena. Oye Luis, una pregunta, espero no sea muy idiota. La verdad es que el segundo argumento me parece potentísimo. En el primero yo me planteo, entiendo que de la manera que habéis, lo voy a decir de manera muy coloquial para que se pueda entender la mayoría de gente,
yo entiendo que ese viaje al futuro, como tú decías, cuando vamos hacia atrás, lo que hacemos es, tomando secuencias de hoy en día, la computadora, los algoritmos lo que hacen es probabilísticamente ver de dónde podría venir un ancestro cada vez más común, hasta que tenemos el famoso Luca, como si fuéramos a buscar a Luca, algo así, entonces vamos para atrás. Pero claro, en ese jugar hacia atrás entiendo que, y esto es una pregunta,
entiendo pero en realidad es una pregunta, ¿se aplica una presión selectiva? Es decir, en ese ir hacia atrás podemos estar olvidando o de alguna manera no pudiendo meterle la presión selectiva que de alguna manera alimenta, la gasolina de la evolución por selección natural y por eso a veces tenemos un poco de deriva. Y en tal caso, ¿cómo se corrige para eso que debe ser tan abstracto y tan difícil?
Pues esa es una buenísima pregunta que nosotros no podemos aplicar. El ordenador no puede aplicar esta presión selectiva. El ordenador lo único que nos puede hacer es, ¿cuáles son la sucesión de aminoácidos que encajaría con las proteínas que conocemos hoy en día? Lo encajaría tras las modificaciones correspondientes. Entonces, el cómo, el camino que ha seguido esa supuesta proteína ancestral
para llegar a la actual necesariamente ha sido gobernado por la evolución y por la selección natural. Ahora ese camino pues no lo sabemos con exactitud. No podemos, porque no podemos poner esa presión selectiva en los algoritmos. Lo que sabemos es que esa secuencia que cuando te la miras dices pues esto no se parece en nada a una núclea actual. Lo que sabemos es que nos dice el ordenador, y esto es un modelo probabilístico,
modelo estadístico, nos dice, esta es la secuencia más probable que debió existir en esta edad y que es compatible con la variabilidad actual. Y repito, no tenemos idea de comprobar, no tenemos manera de comprobar que eso es así. No podemos saber, pero lo que sí podemos saber es que esa proteína es una de las que podría haber existido y podría haber sido la madre que dio lugar a toda la variabilidad actual.
Yo entiendo que esto es una de las limitaciones del estudio porque evidentemente son estimaciones probabilísticas. Pero en estos momentos, mientras la química no nos permita pues recuperar ADN más antiguo de dos millones de años, pues es la única manera que tenemos de hacerlo. Cuando la química nos lo permita, me parece el salto que han hecho estos investigadores descubriendo ADN que está adherido a los minerales, a las arcillas de Groenlandia,
me parece espectacular, pero claro, se han quedado en dos millones y se han quedado en fragmentitos de ADN muy pequeños que son los que han resistido el paso del tiempo. Entonces, ahora mismo se me ocurre o se me antoja bastante complicado ir más allá de dos millones, pero bueno, para eso está la ciencia. Basta que digamos que no es posible encontrar ADN más antiguo de dos millones para que en dos semanas alguien nos diga, pues mira, aquí tienes un ADN de 10 millones
y a partir de ahí, pues yo qué sé. Entonces, mientras no tengamos evidencias directas, pues una manera válida, pero también reconociendo las limitaciones, como estamos haciendo ahora, pues es acudir a estos modelos probabilísticos. Oye, Lluís, ¿qué siguiente cosa o cómo os gustaría proseguir con esta línea? Claro, vamos a ver. Pues solamente, y hay unas comillas enormes, solamente hemos comprobado
que estas casas ancestrales funcionan en células humanas, lo cual es un salto, para mí conceptualmente es muy importante, pero en el fondo, pues lo único que hemos hecho es dársela a unas células en cultivo y ver cómo cortarían las casas actuales que estamos usando. Eso está muy bien, pero eso dista mucho de las aplicaciones reales. Las aplicaciones reales, los sistemas CRISPR tienen que interaccionar
con sistemas más complejos. Entonces, el siguiente paso es saltar desde las células en cultivo a un modelo un poquito más complejo, por ejemplo, un modelo animal, por ejemplo, un ratón. Ya estamos probando estas proteínas en modelos animales, que pueden ser tanto ratones como peces cebra, o nuestros colegas de plantas también probablemente las probarán en plantas, de tal manera que podamos ver cómo funcionan en un modelo real.
Porque no tenemos que olvidar que uno de los talones de Aquiles de los sistemas CRISPR-Cas, al menos de los sistemas CRISPR-Cas sin variaciones, los originales, es que más allá de cortar donde tienen que cortar, a veces pueden cortar en genes parecidos, que no son exactamente los que tú quisieras modificar, y esto puede resultar en consecuencias imprevisibles. Entonces, no tenemos claro cuál va a ser su comportamiento real
cuando estos los pongamos a trabajar. En definitiva, tenemos que investigar su seguridad y su eficacia, antes de incluso plantearnos cualquier traslación a ensayos clínicos o a la realidad. Hay que recordar que todo lo que ocurre con los sistemas CRISPR va a velocidad supersónica, supersónica desde el punto de vista de la biomedicina. Por ejemplo, este año, me refiero al 2022, el año que acaba de terminar,
acaba con una niña con una leucemia bastante agresiva, que era médicamente intratable y que estaba desahuciada, y que gracias a una evolución de los sistemas CRISPR, los llamados editores de bases, se salva. Se salva y ha logrado sobrevivir. Este tratamiento de estos editores de bases que se hace en 2022 es gracias a una herramienta, a una variación de las CRISPR, que son los editores de bases, que la primera vez que se publica es 2016.
Son seis años los que han pasado entre una propuesta de investigación básica y el usarlo directamente. Por el medio ha habido todos los pasos necesarios de células, de animales, de validación, y esto, en definitiva, son parte de los ensayos clínicos. Entonces, en eso estamos. Se trata de acercarnos cada vez más a una situación actual y comprobar, pero no ya teóricamente, sino en la práctica, que nuestra propuesta de que estas nucleasas son lo suficientemente distintas
para que nuestro sistema inmune no las reconozca como parte de las proteínas contra las cuales tiene montado un sistema de ataque, un sistema de respuesta. Pues esto es lo que tenemos que hacer. Qué interesante. Lluís, enhorabuena y muchísimas gracias. Gracias a ti siempre, Lluís, por estar pendiente de estos avances científicos y ayudarnos a contarlo a todo el mundo.


